文件格式
- 为了确保在Win和Linux系统下均可使用,确保数据和系谱保存格式为utf-8。
- 育种数据分析,主要用到2个文件,系谱文件和数据文件。如果开展基因组选择,会使用基于分子标记信息的G或者H矩阵或者逆矩阵文件。在ASReml中,通常以稀疏矩阵格式提供(三列)逆矩阵文件。
- 系谱文件和数据文件,通常保存为.csv文本格式。csv格式就是逗号分隔的文本格式。具体保存方法,在excel中输入数据后,点击保存,选择csv格式。通常在中文Win下excel生成的csv文件是GBK格式,需要用Ultraedit或者Notepad++重新保存为utf-8格式。
- 数据文件名表示方式:性状前缀+世代名称Data.csv,譬如中国对虾2015年生长测试数据文件,可以用g2015G11Data.csv表示。
- 系谱文件名表示方式:性状前缀++世代名称Ped.csv,譬如中国对虾2015年系谱文件,可以用g2015G11Ped.csv表示。
- 如果是基于分子亲缘关系逆矩阵分析,通常扩展名为giv文件,每一列通过空格或者制表符分割。
通用规则
- 数据按照行保存,每一行是一个个体的记录。每一列表示不同的变量,如个体编号,父本编号,性别,出生年份等信息。
- 第一行一般是表头,表示该列字段的意义。譬如第一列是个体编号,通常在第一行用AnimalID来表示。第二列是父本编号,通常用SireID表示。
- 约定俗成的规定,表头一般用英文字符如AnimalID等来表示,因为某些统计分析或者数据处理软件软件不兼容中文。
- 数据文件中,缺失的数据元素,用NA表示。譬如,如果看不出某只虾的性别,用NA代替。如果某只虾的性别鉴定不出来,也用NA表示。
- 数据文件中,性状关联字段,如收获体重、标记时体重等数值字段,务必请检查数据范围,修正明显超出范围的错误值。譬如收获体重均值为10g,如果某只虾的体重为300g、1000g,明显是输入错误;如果存在0.01g,很大概率也是输入错误。
- 对于性状、测试池等分类字段,可以利用Excel中的筛选功能,查看每个字段有多少个水平。譬如性别列,如果出现Male、male、 Female、F、female等,需要统一修正为仅包括两种性别。
- 当用Excel整理数据时,一定要确保单元格中不能为空,如果没有值,用NA填充。系谱文件例外,见下文。
- 如果用ASReml软件进行遗传评估,生成的扩展名为.ass的数据汇总文件务必要首先查看,核对数据是否准确。
- 推荐利用R包DataExplorer包对待分析数据进行检查。
系谱文件
- 系谱文件有三个字段,分别为个体编号、父本编号和母本编号;
- 系谱文件个体数据要按照规则排序:一个个体的父母本系谱信息必须出现在该个体前边;
- 如果个体的父本和母本未知,用0代替。父本和母本要么都为0,如果只知道已授精的母本,父本也要编一个号码;
- 如果系谱文件和数据文件共用一个文件,那么文件的前三列将是个体编号、父本编号和母本编号;
- 如果父本编号和母本编号两列中的个体没有在个体编号列中出现,将会默认为奠基者个体。
- 在第四列加入个体的性别是一个好的建议。
- 可以利用optiSel、visPedigree等R包实现对系谱文件的整理
- visPedigree包的中文安装和使用说明
文件保存
在数据分析过程中会产生大量的文件,为了保证数据分析的可靠性,请务必使用以下文件夹层次保存文件:
PWS2010G01 #育种项目顶层文件夹
—–Data
——–Input #保存原始的系谱和数据文件
——–Output #保存R脚本清洗后的数据和系谱等文件
—–ASReml #保存as文件和ASReml生成的输出文件
—–R #保存清洗数据的R脚本文件
—–BLUPF90 #生成H矩阵的BLUPF90脚本
如果使用ASReml分析数据,as文件中务必使用!OUTFOLDER限定符将不同Path的内容输出到单独文件夹中。关于R文件的编码格式,请参考这里。
数据处理实例
knitr::opts_chunk$set(echo = TRUE)
rm(list=ls()) #清空已存在的各种对象和变量
library(data.table)
library(visPedigree)
library(DataExplorer)1.读取系谱和数据文件
数据文件和系谱文件可以点击下载。
运行R代码前的注意事项和Rstudio编辑器配置和运行等,参见另外一个blog。
下载后,跟R代码放在同一目录下,如果不打算修改下述代码,那么把数据和系谱文件放在一个新建目录datasets下。datasets文件夹和R脚本文件在同一个目录下。
gPed_dt <-
fread(
file = "datasets/g2016G0to2017G1Ped.csv",
sep = ",",
stringsAsFactors = FALSE,
header = TRUE
)
gData_dt <-
fread(
file = "datasets/g2016G0to2017G1Data.csv",
sep = ",",
stringsAsFactors = FALSE,
header = TRUE
)
gPed_dt$Sex[gPed_dt$Sex == "0"] <- NA2. 数据检查和整理
2.1 数据检查
检查数据中分类以及连续性变量的分布,主要是发现是否有异常值。
根据柱状图,可以查看世代、池塘、性别等分类变量的水平数。如果性别有三个,很容易可以查看出来。
根据直方图,可以检查体重、体长、日龄等连续变量的分布,如果有异常值,也非常容易查看。
连续性变量之间的散点图和相关图,用来发现不同变量间的趋势,也是非常有用的工具。
#检查分类变量
plot_bar(gData_dt)## 4 columns ignored with more than 50 categories.
## AnimalID: 11413 categories
## SireID: 90 categories
## DamID: 120 categories
## FamilyID: 120 categories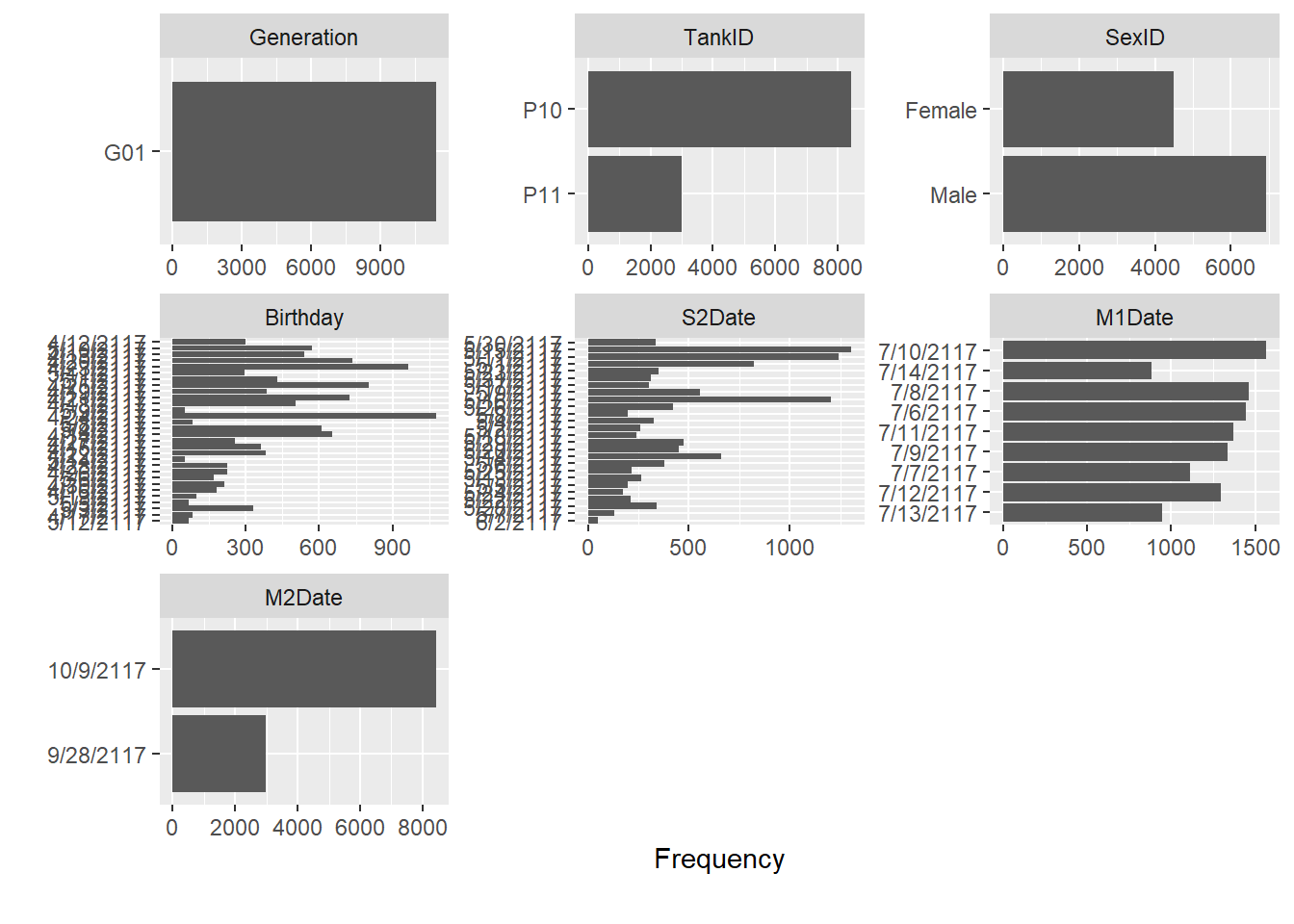
#检查连续性变量
plot_histogram(gData_dt)
#检查连续性变量与收获体重间的散点图
plot_scatterplot(split_columns(gData_dt)$continuous, by = "M2BW")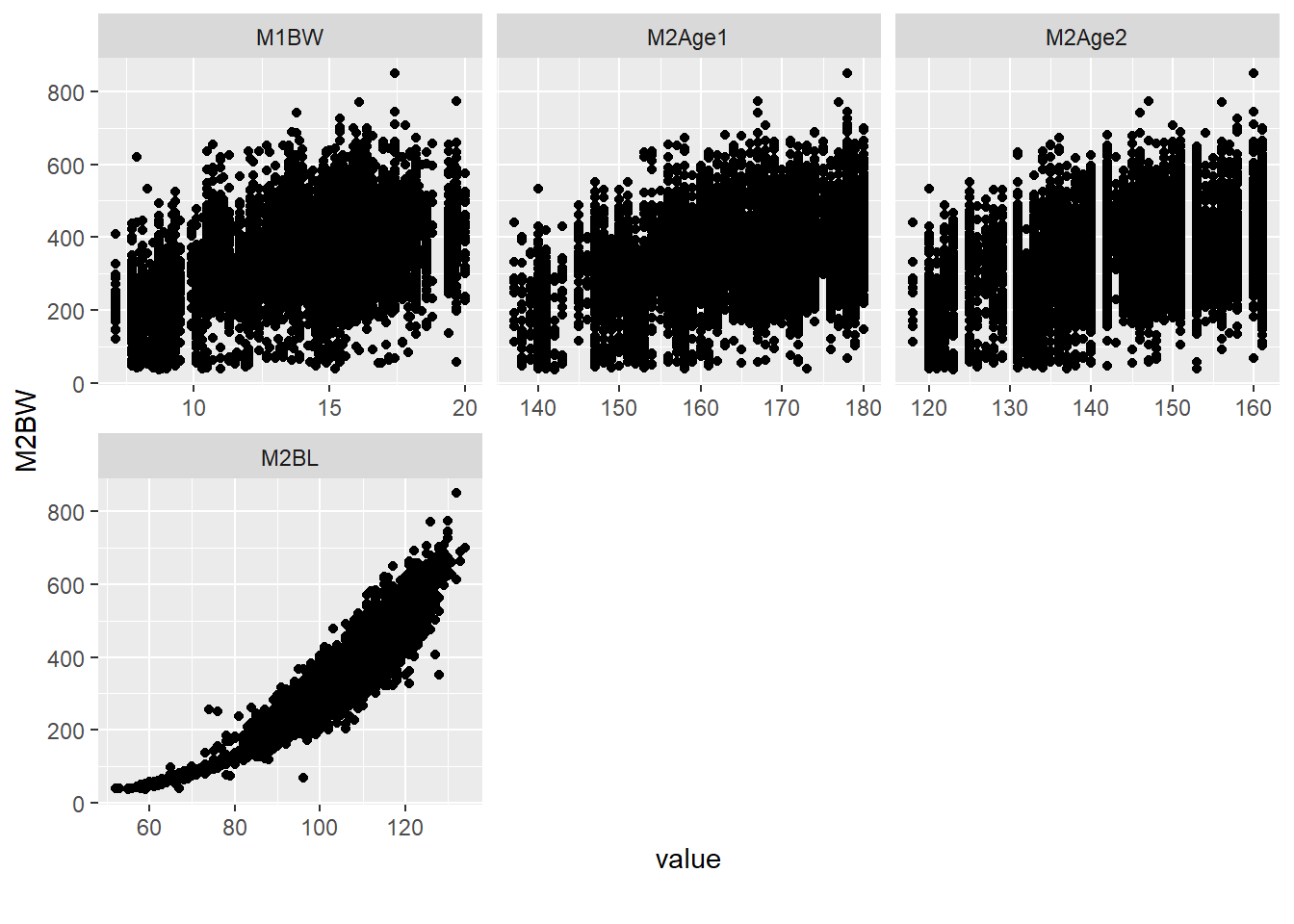
#检查连续性变量间的相关性
plot_correlation(gData_dt, type = "continuous")
2.2 数据清洗
数据分析的需求是经常变动和碎片化的,因此需要掌握熟练的数据清洗技能。数据清洗主流的R包有dplyr和data.table等包。网络上关于二者的比较也有不少。前者的优势是有一个较为完善的数据处理体系,具体参见tidyverse包合集。data.table的优势是特别适合处理大数据,读入、处理和写入的速度非常快。考虑到测序和分型数据都是上G的数据量,推荐利用data.table包。data.table包理解起来,一开始可能有些门槛,不过跟着教程,很快就会熟悉了。我也跟着官方英文教程写了几个学习的blog,供大家参考。百度、知乎上也有不少data.table的使用方法介绍,大家自行参考,具体data.table的使用就不在此赘述了。这里举几个在育种数据分析中用到data.table的例子:
2.2.1 数据提取
(1)提取雄虾数据
有时候,审稿人可能会要求我们单独分析雄虾数据的遗传参数,这就要求我们提取雄虾的数据,利用data.table,实现起来很简单。注意%chin%操作符是data.table单独针对字符类型重构的,速度较快。数值类型,可以直接用>、<等符号。
gData_dt[SexID %chin% c("Female")]## AnimalID SireID DamID Generation
## 1: OXHMB17G010D158 OXHMB16G000A389 OXHMB16G000B754 G01
## 2: OXHMB17G010D261 OXHMB16G000A012 MRTZD16G000F031 G01
## 3: OXHMB17G0101515 OXHMB16G000F297 OXHMB16G000E364 G01
## 4: OXHMB17G0109465 OXHMB16G000A343 MRTZD16G000F026 G01
## 5: OXHMB17G0103104 OXHMB16G000A765 OXHMB16G00X110 G01
## ---
## 4477: OXHMB17G0108813 OXHMB16G000A275 MRTZD16G000F001 G01
## 4478: OXHMB17G0000146 MRTZD16B000M002 MRTZD16B000F002 G01
## 4479: OXHMB17G0110114 MRTZD16G000M052 MRTZD16G000F052 G01
## 4480: OXHMB17G0106486 OXHMB16G000F132 OXHMB16G000F371 G01
## 4481: OXHMB17G010F177 MRTZD16G000M021 OXHMB16G000B013 G01
## FamilyID TankID SexID Birthday S2Date M1Date M1BW
## 1: OXHMB17G0100482 P11 Female 4/22/2117 5/14/2117 7/9/2117 16.4
## 2: OXHMB17G0100891 P11 Female 4/24/2117 5/15/2117 7/10/2117 14.7
## 3: OXHMB17G0100211 P10 Female 4/19/2117 5/9/2117 7/8/2117 15.2
## 4: OXHMB17G0100881 P10 Female 4/18/2117 5/11/2117 7/8/2117 13.6
## 5: OXHMB17G0100323 P10 Female 5/1/2117 5/23/2117 7/12/2117 13.0
## ---
## 4477: OXHMB17G0100831 P10 Female 4/20/2117 5/9/2117 7/8/2117 16.4
## 4478: OXHMB17G0001021 P10 Female 5/10/2117 5/29/2117 7/14/2117 8.7
## 4479: OXHMB17G0100933 P10 Female 4/30/2117 5/22/2117 7/12/2117 11.3
## 4480: OXHMB17G0100611 P10 Female 4/28/2117 5/20/2117 7/11/2117 12.8
## 4481: OXHMB17G0100771 P10 Female 4/14/2117 5/4/2117 7/6/2117 15.4
## M2Date M2Age1 M2Age2 M2BW M2BL
## 1: 9/28/2117 159 137 430 113
## 2: 9/28/2117 157 136 488 117
## 3: 10/9/2117 173 153 337 111
## 4: 10/9/2117 174 151 273 103
## 5: 10/9/2117 161 139 355 109
## ---
## 4477: 10/9/2117 172 153 362 111
## 4478: 10/9/2117 152 133 276 102
## 4479: 10/9/2117 162 140 157 85
## 4480: 10/9/2117 164 142 201 98
## 4481: 10/9/2117 178 158 689 133举一反三,如何提取体重大于700的个体记录集?
gData_dt[M2BW >700]## AnimalID SireID DamID Generation FamilyID
## 1: OXHMB17G010D260 OXHMB16G000F302 OXHMB16G000B783 G01 OXHMB17G0100461
## 2: OXHMB17G010D342 OXHMB16G000A316 OXHMB16G000F439 G01 OXHMB17G0100131
## 3: OXHMB17G010D872 MRTZD16G000M021 OXHMB16G000B013 G01 OXHMB17G0100771
## 4: OXHMB17G010F039 OXHMB16G000C612 OXHMB16G000B679 G01 OXHMB17G0100232
## 5: OXHMB17G010F063 MRTZD16G000M001 OXHMB16G00X112 G01 OXHMB17G0100731
## 6: OXHMB17G010F087 MRTZD16G000M021 OXHMB16G000B013 G01 OXHMB17G0100771
## 7: OXHMB17G010F216 OXHMB16G000A805 OXHMB16G000F485 G01 OXHMB17G0100441
## 8: OXHMB17G010F254 OXHMB16G000C612 OXHMB16G000B679 G01 OXHMB17G0100232
## 9: OXHMB17G010F899 OXHMB16G000C612 OXHMB16G000B679 G01 OXHMB17G0100232
## TankID SexID Birthday S2Date M1Date M1BW M2Date M2Age1 M2Age2 M2BW
## 1: P11 Male 4/14/2117 5/4/2117 7/7/2117 19.7 9/28/2117 167 147 774
## 2: P11 Male 4/13/2117 5/1/2117 7/6/2117 17.8 9/28/2117 168 150 706
## 3: P10 Male 4/14/2117 5/4/2117 7/6/2117 15.4 10/9/2117 178 158 725
## 4: P10 Male 4/14/2117 5/2/2117 7/6/2117 17.4 10/9/2117 178 160 744
## 5: P10 Male 4/25/2117 5/16/2117 7/11/2117 13.8 10/9/2117 167 146 741
## 6: P10 Male 4/14/2117 5/4/2117 7/6/2117 15.4 10/9/2117 178 158 703
## 7: P10 Male 4/15/2117 5/6/2117 7/7/2117 16.1 10/9/2117 177 156 770
## 8: P10 Male 4/14/2117 5/2/2117 7/6/2117 17.4 10/9/2117 178 160 850
## 9: P10 Male 4/14/2117 5/2/2117 7/6/2117 17.4 10/9/2117 178 160 709
## M2BL
## 1: 130
## 2: 125
## 3: 130
## 4: 130
## 5: 130
## 6: 128
## 7: 126
## 8: 132
## 9: 129(2)提取指定家系的雄虾数据
如果想提取特定几个家系,如编号为OXHMB17G0100482、OXHMB17G0100891、OXHMB17G0100211家系的雄虾,如何处理?&是R中的逻辑运算符,表示两边条件都满足时,才返回TRUE。
gData_dt[(FamilyID %chin% c("OXHMB17G0100482", "OXHMB17G0100891", "OXHMB17G0100211")) & (SexID %chin% c("Male"))]## AnimalID SireID DamID Generation FamilyID
## 1: OXHMB17G0105286 OXHMB16G000A389 OXHMB16G000B754 G01 OXHMB17G0100482
## 2: OXHMB17G0101510 OXHMB16G000F297 OXHMB16G000E364 G01 OXHMB17G0100211
## 3: OXHMB17G010A413 OXHMB16G000A012 MRTZD16G000F031 G01 OXHMB17G0100891
## 4: OXHMB17G010A418 OXHMB16G000F297 OXHMB16G000E364 G01 OXHMB17G0100211
## 5: OXHMB17G010A438 OXHMB16G000A012 MRTZD16G000F031 G01 OXHMB17G0100891
## ---
## 191: OXHMB17G0109666 OXHMB16G000A012 MRTZD16G000F031 G01 OXHMB17G0100891
## 192: OXHMB17G0109667 OXHMB16G000A012 MRTZD16G000F031 G01 OXHMB17G0100891
## 193: OXHMB17G0109668 OXHMB16G000A012 MRTZD16G000F031 G01 OXHMB17G0100891
## 194: OXHMB17G0109669 OXHMB16G000A012 MRTZD16G000F031 G01 OXHMB17G0100891
## 195: OXHMB17G0109670 OXHMB16G000A012 MRTZD16G000F031 G01 OXHMB17G0100891
## TankID SexID Birthday S2Date M1Date M1BW M2Date M2Age1 M2Age2
## 1: P11 Male 4/22/2117 5/14/2117 7/9/2117 16.4 9/28/2117 159 137
## 2: P11 Male 4/19/2117 5/9/2117 7/8/2117 16.2 9/28/2117 162 142
## 3: P11 Male 4/24/2117 5/15/2117 7/10/2117 14.7 9/28/2117 157 136
## 4: P11 Male 4/19/2117 5/9/2117 7/8/2117 16.2 9/28/2117 162 142
## 5: P11 Male 4/24/2117 5/15/2117 7/10/2117 14.7 9/28/2117 157 136
## ---
## 191: P10 Male 4/24/2117 5/15/2117 7/10/2117 15.4 10/9/2117 168 147
## 192: P10 Male 4/24/2117 5/15/2117 7/10/2117 15.4 10/9/2117 168 147
## 193: P10 Male 4/24/2117 5/15/2117 7/10/2117 15.4 10/9/2117 168 147
## 194: P10 Male 4/24/2117 5/15/2117 7/10/2117 15.4 10/9/2117 168 147
## 195: P10 Male 4/24/2117 5/15/2117 7/10/2117 15.4 10/9/2117 168 147
## M2BW M2BL
## 1: 169 89
## 2: 417 111
## 3: 522 118
## 4: 604 121
## 5: 515 120
## ---
## 191: 333 103
## 192: 360 109
## 193: 241 102
## 194: 414 109
## 195: 444 113(3)插入新列
譬如给整个数据集加入一个流水号,用于后续数据整理。请注意,:=会直接在原数据集中插入新的一列。如果你不想改变原有数据集,需要提前拷贝原数据集,再插入新列。
gData_new_dt <- copy(gData_dt)
gData_new_dt[,":="(SerialNum=.I)]
gData_new_dt## AnimalID SireID DamID Generation
## 1: OXHMB17G010A338 OXHMB16G000F302 OXHMB16G000B783 G01
## 2: OXHMB17G010A339 OXHMB16G000C364 OXHMB16G00X751 G01
## 3: OXHMB17G010A340 OXHMB16G000C863 OXHMB16G000B274 G01
## 4: OXHMB17G0101354 OXHMB16G000C625 OXHMB16G000F625 G01
## 5: OXHMB17G010A341 OXHMB16G000A300 MRTZD16G000F021 G01
## ---
## 11409: OXHMB17G0104360 OXHMB16G000C514 OXHMB16G000E734 G01
## 11410: OXHMB17G0110468 MRTZD16G000M071 MRTZD16G000F071 G01
## 11411: OXHMB17G0000260 MRTZD16B000M003 MRTZD16B000F003 G01
## 11412: OXHMB17G0000513 MRTZD16B000M005 MRTZD16B000F005 G01
## 11413: OXHMB17G0104470 OXHMB16G000F703 OXHMB16G000F694 G01
## FamilyID TankID SexID Birthday S2Date M1Date M1BW
## 1: OXHMB17G0100461 P11 Male 4/14/2117 5/4/2117 7/7/2117 19.7
## 2: OXHMB17G0100411 P11 Male 4/14/2117 5/1/2117 7/6/2117 16.0
## 3: OXHMB17G0100661 P11 Male 4/24/2117 5/15/2117 7/10/2117 15.2
## 4: OXHMB17G0100201 P11 Male 4/21/2117 5/14/2117 7/9/2117 15.6
## 5: OXHMB17G0100871 P11 Male 4/12/2117 5/1/2117 7/6/2117 18.2
## ---
## 11409: OXHMB17G0100422 P10 Male 5/1/2117 5/23/2117 7/12/2117 14.2
## 11410: OXHMB17G0100971 P10 Male 4/25/2117 5/16/2117 7/11/2117 15.1
## 11411: OXHMB17G0001031 P10 Male 5/10/2117 5/29/2117 7/14/2117 9.1
## 11412: OXHMB17G0001051 P10 Male 5/10/2117 5/30/2117 7/14/2117 7.7
## 11413: OXHMB17G0100433 P10 Male 5/8/2117 5/28/2117 7/13/2117 12.0
## M2Date M2Age1 M2Age2 M2BW M2BL SerialNum
## 1: 9/28/2117 167 147 395 113 1
## 2: 9/28/2117 167 150 408 112 2
## 3: 9/28/2117 157 136 465 120 3
## 4: 9/28/2117 160 137 355 112 4
## 5: 9/28/2117 169 150 581 123 5
## ---
## 11409: 10/9/2117 161 139 214 94 11409
## 11410: 10/9/2117 167 146 156 85 11410
## 11411: 10/9/2117 152 133 142 83 11411
## 11412: 10/9/2117 152 132 174 89 11412
## 11413: 10/9/2117 154 134 50 59 11413.I是data.table中定义的特殊字符,有特殊的含义,这里表示会生成一个从1开始,间隔为1的序列。因此也可以这样实现:
gData_new_dt[,":="(SerialNum=seq(from=1, to=nrow(gData_new_dt), by=1))]
gData_new_dt## AnimalID SireID DamID Generation
## 1: OXHMB17G010A338 OXHMB16G000F302 OXHMB16G000B783 G01
## 2: OXHMB17G010A339 OXHMB16G000C364 OXHMB16G00X751 G01
## 3: OXHMB17G010A340 OXHMB16G000C863 OXHMB16G000B274 G01
## 4: OXHMB17G0101354 OXHMB16G000C625 OXHMB16G000F625 G01
## 5: OXHMB17G010A341 OXHMB16G000A300 MRTZD16G000F021 G01
## ---
## 11409: OXHMB17G0104360 OXHMB16G000C514 OXHMB16G000E734 G01
## 11410: OXHMB17G0110468 MRTZD16G000M071 MRTZD16G000F071 G01
## 11411: OXHMB17G0000260 MRTZD16B000M003 MRTZD16B000F003 G01
## 11412: OXHMB17G0000513 MRTZD16B000M005 MRTZD16B000F005 G01
## 11413: OXHMB17G0104470 OXHMB16G000F703 OXHMB16G000F694 G01
## FamilyID TankID SexID Birthday S2Date M1Date M1BW
## 1: OXHMB17G0100461 P11 Male 4/14/2117 5/4/2117 7/7/2117 19.7
## 2: OXHMB17G0100411 P11 Male 4/14/2117 5/1/2117 7/6/2117 16.0
## 3: OXHMB17G0100661 P11 Male 4/24/2117 5/15/2117 7/10/2117 15.2
## 4: OXHMB17G0100201 P11 Male 4/21/2117 5/14/2117 7/9/2117 15.6
## 5: OXHMB17G0100871 P11 Male 4/12/2117 5/1/2117 7/6/2117 18.2
## ---
## 11409: OXHMB17G0100422 P10 Male 5/1/2117 5/23/2117 7/12/2117 14.2
## 11410: OXHMB17G0100971 P10 Male 4/25/2117 5/16/2117 7/11/2117 15.1
## 11411: OXHMB17G0001031 P10 Male 5/10/2117 5/29/2117 7/14/2117 9.1
## 11412: OXHMB17G0001051 P10 Male 5/10/2117 5/30/2117 7/14/2117 7.7
## 11413: OXHMB17G0100433 P10 Male 5/8/2117 5/28/2117 7/13/2117 12.0
## M2Date M2Age1 M2Age2 M2BW M2BL SerialNum
## 1: 9/28/2117 167 147 395 113 1
## 2: 9/28/2117 167 150 408 112 2
## 3: 9/28/2117 157 136 465 120 3
## 4: 9/28/2117 160 137 355 112 4
## 5: 9/28/2117 169 150 581 123 5
## ---
## 11409: 10/9/2117 161 139 214 94 11409
## 11410: 10/9/2117 167 146 156 85 11410
## 11411: 10/9/2117 152 133 142 83 11411
## 11412: 10/9/2117 152 132 174 89 11412
## 11413: 10/9/2117 154 134 50 59 114132.2.2 数据统计
(1)获得雌雄虾体重的均值
gData_dt[, .(MeanM2BW=mean(M2BW)), by=.(SexID)]## SexID MeanM2BW
## 1: Male 365.4177
## 2: Female 295.4953(2)进一步统计雌雄虾在不同池塘间的体重均值:
gData_dt[, .(MeanM2BW=mean(M2BW)), by=.(SexID, TankID)]## SexID TankID MeanM2BW
## 1: Male P11 350.4550
## 2: Female P11 459.0000
## 3: Female P10 295.4223
## 4: Male P10 376.7736(3)继续增加需求,在统计变量中,加入体长:
gData_dt[, .(MeanM2BW=mean(M2BW), MeanM2BL=mean(M2BL)), by=.(SexID, TankID)]## SexID TankID MeanM2BW MeanM2BL
## 1: Male P11 350.4550 105.5232
## 2: Female P11 459.0000 115.0000
## 3: Female P10 295.4223 103.3449
## 4: Male P10 376.7736 108.2390(3)需求一直在变:进一步统计P11池塘收获了多少尾母虾?
gData_dt[, .(MeanM2BW=mean(M2BW), MeanM2BL=mean(M2BL), N=.N), by=.(TankID, SexID)]## TankID SexID MeanM2BW MeanM2BL N
## 1: P11 Male 350.4550 105.5232 2991
## 2: P11 Female 459.0000 115.0000 2
## 3: P10 Female 295.4223 103.3449 4479
## 4: P10 Male 376.7736 108.2390 3941.N也是data.table中定义的一个特殊符号,表示每个分组的行数。
(4)思考如何满足这样一个需求?提取的数据集,要求每个家系每个池塘母虾数量大于30尾。
gData_dt[, ":="(Size=.N), by=.(FamilyID, TankID, SexID)]
setorder(gData_dt,-Size)
gData_dt[Size >= 30]## AnimalID SireID DamID Generation
## 1: OXHMB17G0100507 OXHMB16G000A414 OXHMB16G00X015 G01
## 2: OXHMB17G010E349 OXHMB16G000A414 OXHMB16G00X015 G01
## 3: OXHMB17G0100508 OXHMB16G000A414 OXHMB16G00X015 G01
## 4: OXHMB17G0100509 OXHMB16G000A414 OXHMB16G00X015 G01
## 5: OXHMB17G010E394 OXHMB16G000A414 OXHMB16G00X015 G01
## ---
## 7903: OXHMB17G0105768 OXHMB16G000F063 OXHMB16G000E752 G01
## 7904: OXHMB17G0102731 OXHMB16G000A116 OXHMB16G000E255 G01
## 7905: OXHMB17G0106033 OXHMB16G000C751 OXHMB16G000E570 G01
## 7906: OXHMB17G0102732 OXHMB16G000A116 OXHMB16G000E255 G01
## 7907: OXHMB17G0102733 OXHMB16G000A116 OXHMB16G000E255 G01
## FamilyID TankID SexID Birthday S2Date M1Date M1BW
## 1: OXHMB17G0100072 P10 Female 4/23/2117 5/15/2117 7/10/2117 17.3
## 2: OXHMB17G0100072 P10 Female 4/23/2117 5/15/2117 7/10/2117 17.3
## 3: OXHMB17G0100072 P10 Female 4/23/2117 5/15/2117 7/10/2117 17.3
## 4: OXHMB17G0100072 P10 Female 4/23/2117 5/15/2117 7/10/2117 17.3
## 5: OXHMB17G0100072 P10 Female 4/23/2117 5/15/2117 7/10/2117 17.3
## ---
## 7903: OXHMB17G0100522 P10 Male 4/21/2117 5/11/2117 7/9/2117 13.9
## 7904: OXHMB17G0100302 P10 Male 4/24/2117 5/18/2117 7/11/2117 13.7
## 7905: OXHMB17G0100561 P10 Male 4/23/2117 5/15/2117 7/10/2117 11.1
## 7906: OXHMB17G0100302 P10 Male 4/24/2117 5/18/2117 7/11/2117 13.7
## 7907: OXHMB17G0100302 P10 Male 4/24/2117 5/18/2117 7/11/2117 13.7
## M2Date M2Age1 M2Age2 M2BW M2BL Size
## 1: 10/9/2117 169 147 294 104 76
## 2: 10/9/2117 169 147 328 107 76
## 3: 10/9/2117 169 147 284 103 76
## 4: 10/9/2117 169 147 306 105 76
## 5: 10/9/2117 169 147 354 109 76
## ---
## 7903: 10/9/2117 171 151 509 118 30
## 7904: 10/9/2117 168 144 285 98 30
## 7905: 10/9/2117 169 147 395 112 30
## 7906: 10/9/2117 168 144 508 120 30
## 7907: 10/9/2117 168 144 420 115 303.预处理系谱、提纯系谱、数字化系谱
visPedigree包中的tidyped()函数,主要有两个功能:
- 包括了自动补全不在系谱ID列中的个体号,以及提纯功能。
- 把与当前数据集中个体没有亲缘关系的系谱个体,全部剔除掉
#预处理系谱,主要是把父母本的系谱设置为0
gPed_tidy_dt <- tidyped(ped = gPed_dt, cand = gData_dt$AnimalID)## Warning in checkped(ped, addgen): In the sire or dam column, Blank, Zero,
## asterisk, or character NA is recognized as a missing parent and is replaced with
## the missing value NA.gPed_tidy_dt## Ind Sire Dam Sex Offspring Year
## 1: OLHBB14B000F001 <NA> <NA> female TRUE 2014
## 2: OLHBB14B000F002 <NA> <NA> female TRUE 2014
## 3: OLHBB14B000F003 <NA> <NA> female TRUE 2014
## 4: OLHBB14B000F004 <NA> <NA> female TRUE 2014
## 5: OLHBB14B000F005 <NA> <NA> female TRUE 2014
## ---
## 11914: OXHMB17G0110820 OLHBC16G000M088 OLHBC16G000F088 male FALSE 2017
## 11915: OXHMB17G0110821 OLHBC16G000M088 OLHBC16G000F088 male FALSE 2017
## 11916: OXHMB17G0110822 OLHBC16G000M088 OLHBC16G000F088 male FALSE 2017
## 11917: OXHMB17G0110823 OLHBC16G000M088 OLHBC16G000F088 male FALSE 2017
## 11918: OXHMB17G0110824 OLHBC16G000M088 OLHBC16G000F088 male FALSE 2017
## Breed Cand Gen IndNum SireNum DamNum
## 1: DP FALSE 1 1 0 0
## 2: DP FALSE 1 2 0 0
## 3: DP FALSE 1 3 0 0
## 4: DP FALSE 1 4 0 0
## 5: DP FALSE 1 5 0 0
## ---
## 11914: unknown TRUE 4 11914 350 332
## 11915: unknown TRUE 4 11915 350 332
## 11916: unknown TRUE 4 11916 350 332
## 11917: unknown TRUE 4 11917 350 332
## 11918: unknown TRUE 4 11918 350 332整理后系谱的包含的个体数,要少于原始系谱。
4.画出个体的系谱树
侧面验证一下提纯后的系谱,是否正确。
从数据集中最后一尾虾,画出系谱图。
这里需要注意的是,visped()函数要求个体的父本和母本,要么双亲独有,要么都没有,不能一个有,一个没有。
实际上可能会存在这样一个情况,像两只已经交尾的母虾,有可能来自同一尾公虾,但是即便是这种情况,也需要给父本一个随机编号,假定他们是各不相同的。
即便是设置为缺失,应该也是这种情况。
one_ped_dt <-
tidyped(ped = gPed_tidy_dt, cand = gData_dt$AnimalID[nrow(gData_dt)])
visped(ped = one_ped_dt)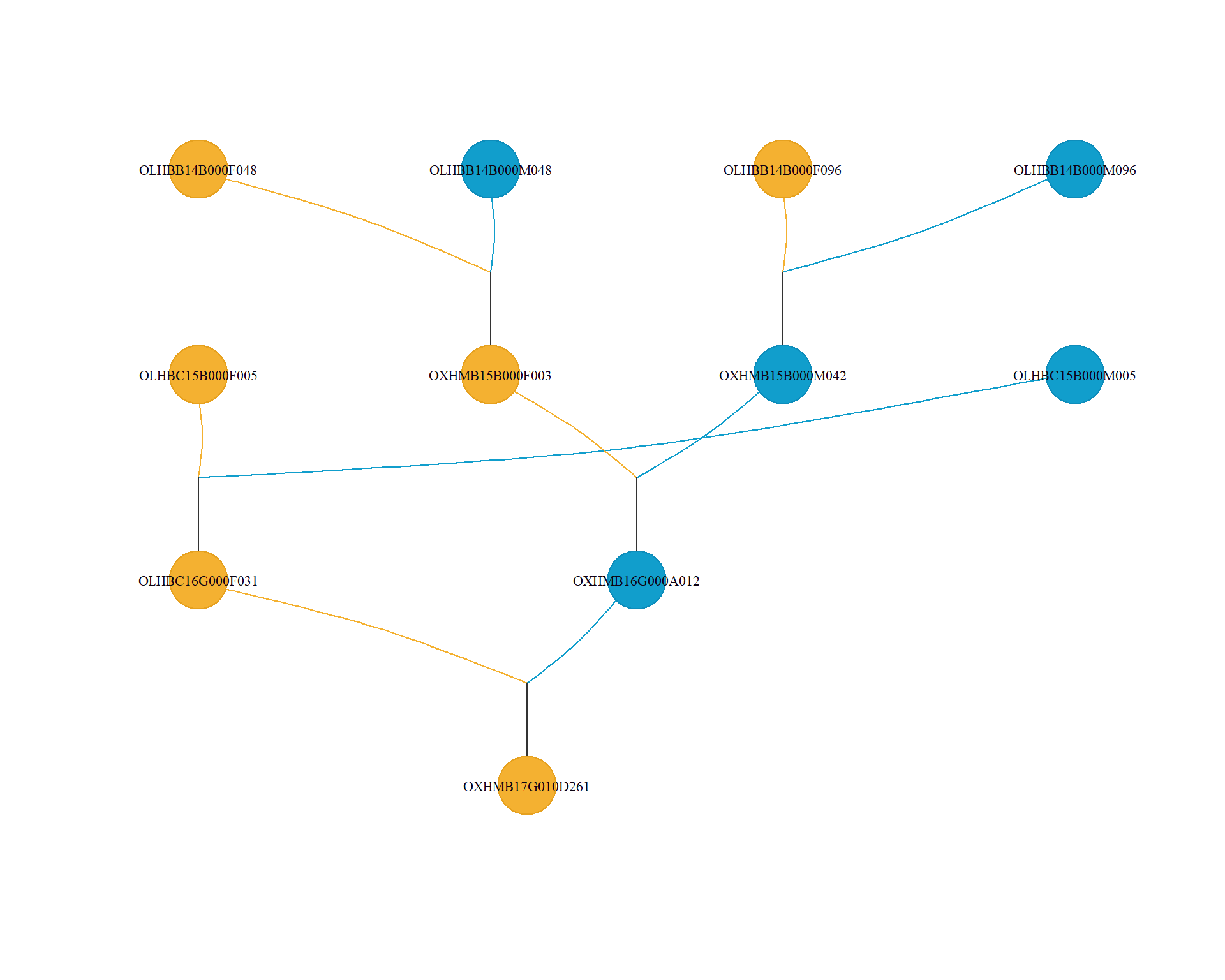
5.输出系谱
输出系谱时,设置缺失的父本和母本编号为0。
fwrite()函数来自data.table包。
gPed_tidy_dt[is.na(Sire), Sire := "0"]
gPed_tidy_dt[is.na(Dam), Dam := "0"]
fwrite(
gPed_tidy_dt,
file = "datasets/g2016G0to2017G1Pedpruned.csv",
sep = ",",
row.names = F,
na = "NA",
quote = FALSE
)