在动物选择育种中,系谱的作用不言而喻。一方面利用系谱信息,可以提高育种值估计和选择的准确度;另外,可以控制近亲交配,避免性状衰退。因此,一个可靠准确的系谱记录对于选择育种是非常重要的。此外,系谱往往通过个体、父本和母本三列形式保存,很难直观的查看个体的祖先和后代信息。因此,系谱的可视化就变得非常重要。在windows平台下,The University of Minnesota杨达教授课题组开发了一个可用于显示个体系谱的pedigraph软件,可以显示很多个体的系谱,功能非常强大,但需要利用参数文件,配置使用。 The University of New England的Brian Kinghorn教授开发了pedigree viewer软件,可以整理、提纯系谱,并且可以在窗口中直观地显示个体的系谱,但如果个体的数量非常多,个体将重叠在一起,系谱显示还需要进一步优化。在R环境下,pedigree,nadiv,optiSel等包均有系谱整理的功能,kinship2, synbreed等包都可以画图,但是一旦系谱包括的个体数量多,画出来的系谱树,个体间重叠非常厉害。
因此利用data.table和igraph两个包处理数据、绘图能力强的特点,开发了visPedigree包,进一步增强了系谱整理的功能,可以设置世代数,追溯提取任意个体的祖先和后代个体;并通过简化系谱和显示系谱缩略图等方法,自动优化系谱树布局、快速显示包括大量个体(每个世代个体数>10000)的系谱。主要内容包括:
1 安装visPedigree包
2 系谱格式说明
3 检查和整理系谱
3.1 概述
3.2 提取指定个体的系谱
3.3 数字化系谱
4 绘制系谱
4.1 绘制简单系谱图
4.2 绘制简化系谱图
4.3 绘制系谱框架图
4.4 应用场景
4.4.1 追踪个体的奠基者组成情况
4.4.2 观察配种方案中不同家系的贡献情况
1 安装visPedigree包
目前visPedigree包还没有发布到cran,但利用devtools包,可以从github上进行安装。
以Rstudio平台为例,如果没有安装devtools包,首先安装,然后加载devtools。
is_installed <- require(devtools)## 载入需要的程辑包:devtools## 载入需要的程辑包:usethisif (!is_installed) {
install.packages("devtools")
suppressPackageStartupMessages(require(devtools))
}从github上安装visPedigree,然后加载visPedigree包。该包基于data.table和igraph包开发,如果这两个包没有安装,也会一并安装。
is_installed <- require(visPedigree)## 载入需要的程辑包:visPedigreeif (!is_installed) {
install_github("luansheng/visPedigree")
suppressPackageStartupMessages(require(visPedigree))
}在国内,直接利用上述方法安装,可能会比较慢,甚至经常会发生安装失败的问题。因此利用devtools::install_local()函数直接从本地安装是一个更好的方法。具体操作如下:
- 访问visPedigree包的github网址。
- 点击页面右侧的Clone or download下拉框,选择Download ZIP,把安装visPedigree包需要的文件下载到本地,保存到一个英文目录下,如
c:/visPedigree。如果是从master分支下载的压缩包,下载后的名字为visPedigree-master.zip。
- 打开Rstudio,切换工作目录到
c:/visPedigree - 利用devtools::install_local()函数进行安装。
setwd("c:/visPedigree")
install_local("visPedigree-master.zip")2 系谱格式说明
系谱数据的前三列顺序必须为个体、父本和母本ID,系谱文件中可以包括其他信息如性别、出生年月、世代等。
个体丢失的亲本可以用0、*、“NA”等表示,推荐用0表示。空格和空值也可表示丢失亲本,但是不推荐使用。个体列如果存在这些值,该行将直接被删除。 其他列如性别中的丢失值,请直接用NA来表示。
如果从文件中读取系谱信息,建议使用data.table包的fread函数。该函数功能非常强大,可以自动识别文本中各种分隔符。
ped_2 <- data.table::fread(file="datasets/ped2.csv",
sep=",",
header = TRUE,
stringsAsFactors = FALSE)
head(ped_2)## ID Sire Dam Sex Cand
## 1: X0YY0500 0 0 Male 0
## 2: X0YY0600 0 0 Male 0
## 3: X0YY0700 0 0 Male 0
## 4: X0YY1200 0 0 Male 0
## 5: X0YX0300 0 0 Male 0
## 6: X0YX0400 0 0 Male 03 检查和整理系谱
3.1 概述
通过tidyped函数可以检查和整理系谱,主要包括:
- 检查系谱是否存在重复个体编号;如果存在,删除重复个体,并给出提示;
- 检查是否存在同时做父本和母本的个体;如果存在,给出提示;
- 检查是否存在系谱循环Pedigree loop,即个体互为祖先和后裔;如果存在,给出提示并停止运行程序;
- 将未包括在个体列的奠基者个体加入到系谱中,并将其双亲设置为丢失值NA;
- 将祖先个体排序在后裔个体前边,进行系谱个体重排序;
- 检查个体的性别,补充丢失的性别信息;
- 生成数字化系谱;
- 提取指定个体、指定世代的祖先和后裔系谱;
visPedigree包中自带多个数据集。可以通过下行命令查看。
data(package="visPedigree")选择simple_ped系谱数据集。它包括四列,前三列为个体、父本和母本,最后一列是性别。其中一些个体的亲本丢失,用0、*、NA等表示,而且奠基者个体没有补充在系谱中。并且一些亲本排在子代的后边。
head(simple_ped)## ID Sire Dam Sex
## 1: J4Y326 J3Y620 J3Y771 male
## 2: J1H419 J0Z938 J0Z167 female
## 3: J2F588 NA J1Z417 female
## 4: J1J576 J0Z938 J0Z843 male
## 5: J1C802 J0Z333 J0C355 male
## 6: J2Z411 J1X971 J1J134 femaletail(simple_ped)## ID Sire Dam Sex
## 1: J1E852 J0Z848 J0Z624 female
## 2: J1H604 J0C583 J0Z380 female
## 3: J5X804 J4Y326 J4E185 female
## 4: J1I438 J0Z990 J0Z808 male
## 5: J2C808 J1I975 J1F266 male
## 6: J1K462 J0C317 J0C450 female# 系谱数据集包括的个体数
nrow(simple_ped)## [1] 31# 查找亲本丢失的个体记录信息
simple_ped[Sire %in% c("0","*","NA",NA) | Dam %in% c("0","*","NA",NA)]## ID Sire Dam Sex
## 1: J2F588 NA J1Z417 female
## 2: J1J858 J0Z060 * female
## 3: J3X697 J2Z903 0 female小测试:将J0Z167雌性个体,设为J2F588个体的父本。然后运行tidyped,将会发现这个问题并给出提示。
x <- data.table::copy(simple_ped)
x[ID == "J2F588",Sire:="J0Z167"]
y <- tidyped(x)## Warning in checkped(ped_inter, addgen): In the sire or dam column, Blank, Zero,
## asterisk, or character NA is recognized as a missing parent and is replaced with
## the missing value NA.## Warning in checkped(ped_inter, addgen): The following individuals are
## simultaneously bisexual.## Warning in checkped(ped_inter, addgen): J0Z167利用tidyped函数整理simple_ped系谱,将丢失的亲本替换为NA,将子代排在亲本前边,在系谱中补充丢失的奠基者个体信息。
tidy_simple_ped <- tidyped(simple_ped)## Warning in checkped(ped_inter, addgen): In the sire or dam column, Blank, Zero,
## asterisk, or character NA is recognized as a missing parent and is replaced with
## the missing value NA.head(tidy_simple_ped)## Ind Sire Dam Sex Gen IndNum SireNum DamNum
## 1: J0C032 <NA> <NA> female 1 1 0 0
## 2: J0C185 <NA> <NA> female 1 2 0 0
## 3: J0C231 <NA> <NA> female 1 3 0 0
## 4: J0C317 <NA> <NA> male 1 4 0 0
## 5: J0C450 <NA> <NA> female 1 5 0 0
## 6: J0C561 <NA> <NA> male 1 6 0 0tail(tidy_simple_ped)## Ind Sire Dam Sex Gen IndNum SireNum DamNum
## 1: J1C802 J0Z333 J0C355 male 5 54 47 46
## 2: J4E185 J3L886 J3X697 female 5 55 48 49
## 3: J4Y326 J3Y620 J3Y771 male 5 56 50 51
## 4: J1C929 J0Z511 J0Z444 male 6 57 53 52
## 5: J2Y434 J1C802 J1H419 female 6 58 54 28
## 6: J5X804 J4Y326 J4E185 female 6 59 56 55nrow(tidy_simple_ped)## [1] 59整理后的系谱数据集,补充了奠基者个体及其性别信息,并且将亲本排序在后代前边,个体数量变为由31增加为59。个体、父本和母本的列名字重新命名为Ind、Sire和Dam。丢失的亲本统一替换为NA,tidyped函数运行后会有相应提示。tidyped函数整理后的系谱数据集默认增加了Gen和IndNum、SireNum和DamNum四列。如果simple_ped没有包括Sex列,也会增加该列。
把addgen和addnum参数设置为FALSE,整理后的系谱文件将不返回Gen和IndNum、SireNum和DamNum四列。
tidy_simple_ped_no_gen_num <- tidyped(simple_ped,addgen = FALSE,addnum = FALSE)## Warning in checkped(ped_inter, addgen): In the sire or dam column, Blank, Zero,
## asterisk, or character NA is recognized as a missing parent and is replaced with
## the missing value NA.head(tidy_simple_ped_no_gen_num)## Ind Sire Dam Sex
## 1: J0Z333 <NA> <NA> male
## 2: J0Z511 <NA> <NA> male
## 3: J0Z664 <NA> <NA> male
## 4: J0Z848 <NA> <NA> male
## 5: J0Z475 <NA> <NA> male
## 6: J0Z938 <NA> <NA> male整理完毕后的系谱,可以利用data.table包的fwrite函数保存为文件,供ASReml等遗传评估软件使用。 保存为系谱文件时,丢失的亲本统一用0代替。
saved_ped <- data.table::copy(tidy_simple_ped)
saved_ped[is.na(Sire),Sire:="0"]
saved_ped[is.na(Dam),Dam:="0"]
data.table::fwrite(x=saved_ped,file = "tidysimpleped.csv",sep=",",quote = FALSE)
head(saved_ped)3.2 提取指定个体的系谱
设置cand参数,可以追溯指定个体的系谱。并且会在返回的数据集中增加新的一列Cand。TRUE表示该个体为cand个体。
tidy_simple_ped_J5X804_ancestors <- tidyped(ped=tidy_simple_ped_no_gen_num,cand="J5X804")
tail(tidy_simple_ped_J5X804_ancestors)## Ind Sire Dam Sex Cand Gen IndNum SireNum DamNum
## 1: J3X697 J2Z903 <NA> female FALSE 4 45 43 0
## 2: J3Y620 J2C161 J2Z411 male FALSE 4 46 37 42
## 3: J3Y771 J2G465 J2X544 female FALSE 4 47 40 41
## 4: J4E185 J3L886 J3X697 female FALSE 5 48 44 45
## 5: J4Y326 J3Y620 J3Y771 male FALSE 5 49 46 47
## 6: J5X804 J4Y326 J4E185 female TRUE 6 50 49 48默认追溯个体至奠基者祖先的系谱。如果只想追溯几代,可以设置tracegen参数。
tidy_simple_ped_J5X804_ancestors_2 <- tidyped(ped=tidy_simple_ped_no_gen_num,cand="J5X804",tracegen = 2)
print(tidy_simple_ped_J5X804_ancestors_2)## Ind Sire Dam Sex Cand Gen IndNum SireNum DamNum
## 1: J3L886 <NA> <NA> male FALSE 1 1 0 0
## 2: J3X697 <NA> <NA> female FALSE 1 2 0 0
## 3: J3Y620 <NA> <NA> male FALSE 1 3 0 0
## 4: J3Y771 <NA> <NA> female FALSE 1 4 0 0
## 5: J4E185 J3L886 J3X697 female FALSE 2 5 1 2
## 6: J4Y326 J3Y620 J3Y771 male FALSE 2 6 3 4
## 7: J5X804 J4Y326 J4E185 female TRUE 3 7 6 5上述代码将追溯个体J5X804两个世代。
如果对个体的后代感兴趣,可以通过设置trace参数来实现。
trace参数有三个选项:
- “up”-追溯至祖先个体;
- “down”-追溯至后裔个体;
- “all”-同时追溯祖先和后裔个体。
tidy_simple_ped_J0Z990_offspring <- tidyped(ped=tidy_simple_ped_no_gen_num,can="J0Z990",trace="down")
print(tidy_simple_ped_J0Z990_offspring)## Ind Sire Dam Sex Cand Gen IndNum SireNum DamNum
## 1: J0Z990 <NA> <NA> male TRUE 1 1 0 0
## 2: J1I438 J0Z990 <NA> male FALSE 2 2 1 0
## 3: J2G465 J1I438 <NA> male FALSE 3 3 2 0
## 4: J3Y771 J2G465 <NA> female FALSE 4 4 3 0
## 5: J4Y326 <NA> J3Y771 male FALSE 5 5 0 4
## 6: J5X804 J4Y326 <NA> female FALSE 6 6 5 0追溯J0Z990的子代,可以发现共计有5个后裔个体。
3.3 数字化系谱
一些程序,会要求提供数字系谱进行遗传评估。在计算加性遗传相关矩阵时,也要求个体连续编号。
tidyped函数默认会增加一个三列(IndNum、SireNum和DamNum)的数字系谱。如果不需要,可以设置addnum=FALSE将该功能关闭。
4 绘制系谱
利用visped函数,可以绘制多代系谱图。系谱图可以在R、Rstudio默认图形设备显示,并且可以保存在pdf文件中。保存在pdf文件中的系谱图是矢量图,更为清晰、可辨认,个体间不会发生重叠现象。当系谱中个体的数量多,个体的ID编号较长时,适合保存在pdf文件中。如果很多个体为全同胞个体,譬如水产动物的核心育种群体,可以对系谱进行简化显示,这样即使每代个体数量超过10000,也可以非常快的绘制出系谱图。如果每个世代包括的个体数量太多,且个体的ID太长,受限于pdf文件的宽度(最大为200英寸),无法绘制系谱图时,将不显示个体的ID号,只显示一个系谱的缩略图,用于帮助育种者快速浏览核心群体的构建过程,查看是否有外血引入。
重要提示:强烈推荐在整理系谱时,指定cand参数。根据cand参数指定的个体提纯的系谱,个体所属世代的推断更加准确,个体在绘制出的系谱图中所在的位置将更为合理。
绘制一个小系谱图,高清矢量图见pdf文件:
tidy_small_ped <-
tidyped(ped = small_ped,
cand = c("Y","Z1","Z2"))
visped(tidy_small_ped, compact = TRUE, file="doc/smallped.pdf")## The vector drawing of the pedigree is saved in the C:/Users/luansheng/OneDrive/HugoMini/content/post/doc/smallped.pdf file## The cex for individual label is 0.7.## Please decrease or increase the value of the parameter cex if the label's width is longer or shorter than that of the circle or square in the graph.
在系谱图中,使用了2种形状和3种颜色来表示各种信息。圆形代表个体;正方形代表简化后的全同胞个体,内部数字为全同胞个体数。暗天蓝色表示雄性,暗金黄色表示雌性,暗橄榄绿表示性别未知。暗天蓝色圆形,内部为字母R,表示雄性个体;暗金黄色圆形,内部为字母W,表示雌性个体;暗橄榄绿色圆形,内部为字母Y,表示性别未知个体;暗橄榄绿色方形,内部为数字2,表示2个全同胞个体,性别未知;个体系谱图上顶端为祖先个体,底部为后裔个体。亲本和子代之间,通过一个点进行连接,子代到中间节点的连线均为深灰色,中间节点连接到父本和母本的线,与父母本各自的颜色相同。
4.1 绘制简单系谱图
绘制整理后的simple_ped系谱图,在R或者Rstudio默认图形设备显示。整理系谱数据集时,需要将tidyped函数的addgen和addnum参数设置为TRUE。
visped(tidy_simple_ped)## The cex for individual label is 0.7.## Please decrease or increase the value of the parameter cex if the label's width is longer or shorter than that of the circle or square in the graph.## It is recommended that the pedigree graph is saved in the pdf file using the parameter file## The graph in the pdf file is a vector drawing: shapes, labels and lines are legible; shapes and labels isn't overlapped.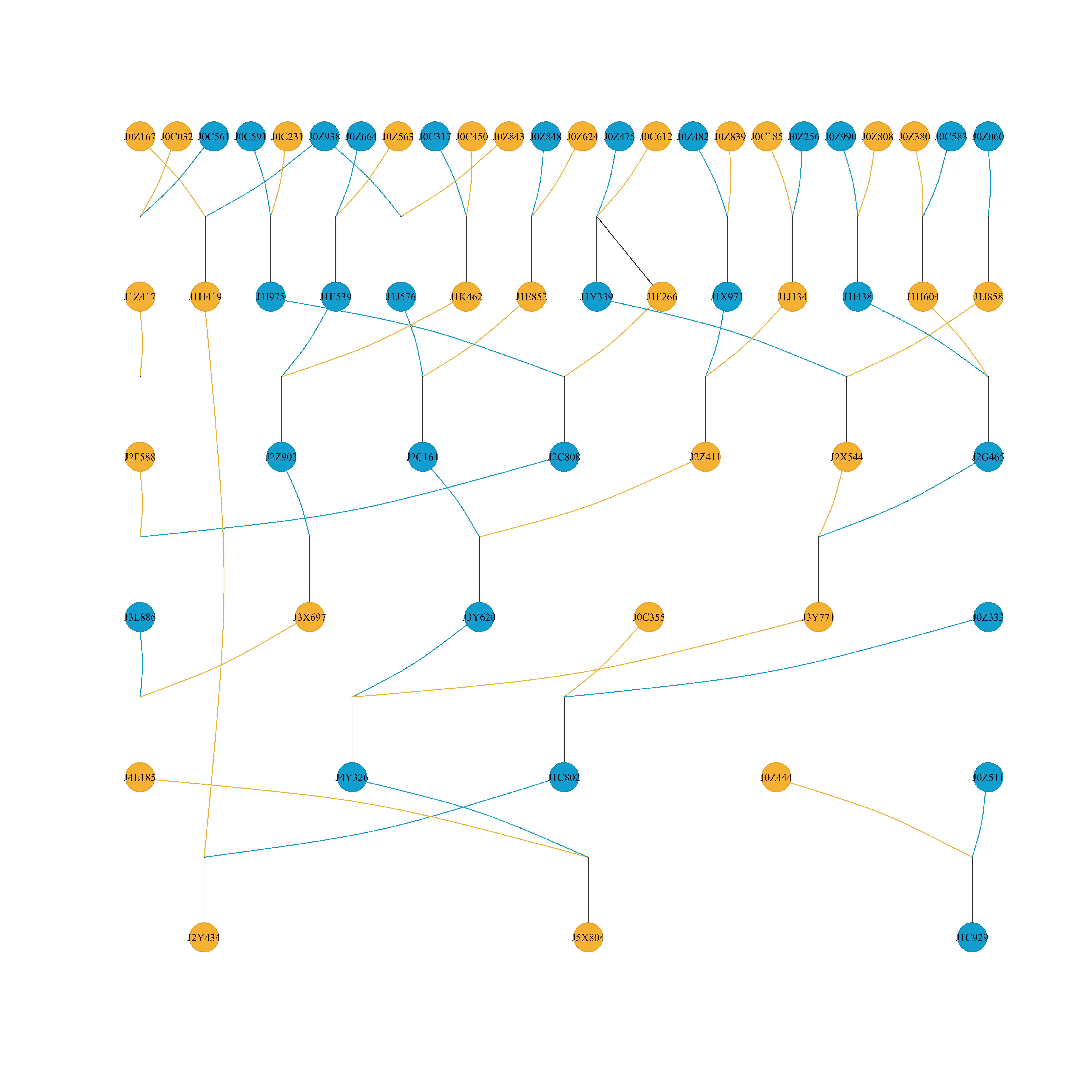
通常在Rstudio的Plots面板上显示的图片,清晰度较差,如果个体数量多,受限于默认的图片显示尺寸,个体ID相互间也会发生重叠。将系谱图存为pdf文件可以有效的避免上述问题,获得高质量的矢量图。设置showgraph = FALSE,将不在默认图形设备上输出图形。
suppressMessages(visped(tidy_simple_ped, showgraph = FALSE, file="doc/simpleped.pdf"))打开simpleped.pdf文件,会看到高清晰度的系谱。
4.2 绘制简化系谱图
对包自带的deep_ped进行整理,然后绘制系谱图
visped(tidyped(deep_ped)) Too many individuals (>=3362) in one generation!!! Two choices:
1. Removing full-sib individuals using the parameter compact = TRUE; or,
2. Visualizing all nodes without labels using the parameter outline = TRUE.
Rerun visped() function!函数提示,某个世代包括的个体数量太多,无法绘制系谱图。建议利用compact或者outline参数来简化系谱图。
首先尝试一下compact参数,并且输出到deepped1.pdf文件中。在默认图形设备上输出的图片,由于个体数量太多,受限于默认设置的图片尺寸不够大,因此重叠问题严重。
visped(tidyped(deep_ped),compact = TRUE, showgraph=TRUE, file="doc/deepped1.pdf")## Warning in checkped(ped_inter, addgen): In the sire or dam column, Blank, Zero,
## asterisk, or character NA is recognized as a missing parent and is replaced with
## the missing value NA.## Warning in checkped(ped_inter, addgen): Blank and NA are recoded as a missing
## sex in the Sex column of the pedigree.## The vector drawing of the pedigree is saved in the C:/Users/luansheng/OneDrive/HugoMini/content/post/doc/deepped1.pdf file## The cex for individual label is 0.525.## Please decrease or increase the value of the parameter cex if the label's width is longer or shorter than that of the circle or square in the graph. 打开deepped1.pdf文件,查看高清的矢量系谱图。最低端个体大部分为正方形,内部的数字是简化后不同家系的全同胞雄性和雌性个体数。如果感觉个体的ID长度小于方形或者圆形,不是很匹配,可以将visped函数给出的cex参数值,进一步变大。cex用来控制图中个体label(ID)的大小,cex越大,个体ID越大,反之亦然。cex取值范围一般为0-1,也可以大于1,每次调整,以0.1作为一个单位。
打开deepped1.pdf文件,查看高清的矢量系谱图。最低端个体大部分为正方形,内部的数字是简化后不同家系的全同胞雄性和雌性个体数。如果感觉个体的ID长度小于方形或者圆形,不是很匹配,可以将visped函数给出的cex参数值,进一步变大。cex用来控制图中个体label(ID)的大小,cex越大,个体ID越大,反之亦然。cex取值范围一般为0-1,也可以大于1,每次调整,以0.1作为一个单位。
visped(tidyped(deep_ped),compact = TRUE, cex=0.83, showgraph = FALSE, file="doc/deepped2.pdf")## Warning in checkped(ped_inter, addgen): In the sire or dam column, Blank, Zero,
## asterisk, or character NA is recognized as a missing parent and is replaced with
## the missing value NA.## Warning in checkped(ped_inter, addgen): Blank and NA are recoded as a missing
## sex in the Sex column of the pedigree.## The vector drawing of the pedigree is saved in the C:/Users/luansheng/OneDrive/HugoMini/content/post/doc/deepped2.pdf file## The cex for individual label is 0.83.## Please decrease or increase the value of the parameter cex if the label's width is longer or shorter than that of the circle or square in the graph.再打开deepped2.pdf文件,查看高清的矢量系谱图,ID与图形的匹配度更高。如果感觉不合适,可以继续调整cex。
4.3 绘制系谱框架图
设置outline=TRUE绘制系谱的框架图,该图将不会显示系谱个体的ID号。当系谱包括的个体数量特别多时,框架图将非常有效。
通过这个图,可以更为清晰的发现在某些世代有外血群体引入。生成的pdf文件请点击这里。
suppressMessages(visped(tidyped(deep_ped),compact = TRUE, outline=TRUE, showgraph = TRUE, file="doc/deepped3.pdf"))## Warning in checkped(ped_inter, addgen): In the sire or dam column, Blank, Zero,
## asterisk, or character NA is recognized as a missing parent and is replaced with
## the missing value NA.## Warning in checkped(ped_inter, addgen): Blank and NA are recoded as a missing
## sex in the Sex column of the pedigree.
绘制包中另外一个系谱数据集,其中包括了大量的全同胞个体,只提取2007年出生个体及其祖先的系谱。pdf系谱图见这里。
cand_2007_G8_labels <- big_family_size_ped[(Year == 2007) & (substr(Ind,1,2) == "G8"),Ind]
cand_2007_G8_tidy_ped <- tidyped(big_family_size_ped,cand=cand_2007_G8_labels)## Warning in checkped(ped_inter, addgen): In the sire or dam column, Blank, Zero,
## asterisk, or character NA is recognized as a missing parent and is replaced with
## the missing value NA.# 利用suppressMessages,禁止输出提示信息
suppressMessages(visped(cand_2007_G8_tidy_ped,compact = TRUE, outline=TRUE, showgraph = TRUE, file="doc/bigfullsibped.pdf"))
4.4 应用场景
4.4.1 追踪个体的奠基者情况
选择育种的过程,实际上就是将分散在多个奠基者个体中的有利基因,通过连续不断的多代交配设计,富集到后代个体中的一个过程。它背后的支持理论,就是大家熟知的微效多基因假说。
我们选择deep_ped数据集中的一尾个体“K110550H”,可视化它的系谱。pdf系谱图见这里。
suppressWarnings(K110550H_ped <- tidyped(deep_ped,cand="K110550H"))
suppressMessages(visped(K110550H_ped,showgraph = TRUE,file="doc/K110550Hped.pdf")) 从上图中可以看出,K110550H个体的奠基者个体(没有亲本)数量为71个,这表示该个体富集了分散在上述奠基者个体的多个有利基因,因此育种目标性状才会表现更加优良。
从上图中可以看出,K110550H个体的奠基者个体(没有亲本)数量为71个,这表示该个体富集了分散在上述奠基者个体的多个有利基因,因此育种目标性状才会表现更加优良。
4.4.2 观察配种方案中不同家系贡献个体情况
当利用最佳贡献理论进行优化配种时,每个家系贡献的个体数量是不一致,综合选择指数高的家系,贡献更多的个体。通过可视化系谱,可以直观地查看不同家系的贡献比例。
下边代码,将展示核心育种群2007年出生的106个家系的亲本组成情况。注意在tidyped函数中设置tracegen=2,只展示父母和祖父母两个世代。
suppressWarnings(
cand_2007_G8_tidy_ped_ancestor_2 <-
tidyped(
big_family_size_ped,
cand = cand_2007_G8_labels,
trace = "up",
tracegen = 2)
)
sire_label <-
unique(cand_2007_G8_tidy_ped_ancestor_2[Ind %in% cand_2007_G8_labels,
Sire])
dam_label <-
unique(cand_2007_G8_tidy_ped_ancestor_2[Ind %in% cand_2007_G8_labels,
Dam])
sire_dam_label <- unique(c(sire_label, dam_label))
sire_dam_label <- sire_dam_label[!is.na(sire_dam_label)]
sire_dam_ped <-
cand_2007_G8_tidy_ped_ancestor_2[Ind %in% sire_dam_label]
sire_dam_ped <- sire_dam_ped[, FamilyID := paste(Sire, Dam, sep = "")]
family_size <- sire_dam_ped[, .N, by = c("FamilyID")]
fullsib_family_label <- unique(sire_dam_ped$FamilyID)
suppressMessages(
visped(
cand_2007_G8_tidy_ped_ancestor_2,
compact = TRUE,
outline = TRUE,
showgraph = TRUE
)
) 上图中,底部为核心群106个家系,中间为其父母本个体,顶部为祖父母个体。可以看出,父母本共计由80个父本、106个母本组成。从上图中可以看出,父母本来自54个全同胞家系,由于采用了最佳贡献理论配种,其中25个父母本来自两个全同胞家系,约占父母本总数的13.44%。
上图中,底部为核心群106个家系,中间为其父母本个体,顶部为祖父母个体。可以看出,父母本共计由80个父本、106个母本组成。从上图中可以看出,父母本来自54个全同胞家系,由于采用了最佳贡献理论配种,其中25个父母本来自两个全同胞家系,约占父母本总数的13.44%。